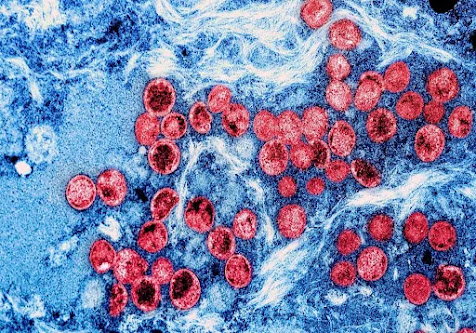
Bukti A.2 Garis Keturunan Cacar Monyet Bermutasi
Dalam penelitian terbaru yang diposting ke preprint server bioRxiv*, tim peneliti dari India menganalisis seluruh urutan genom virus monkeypox yang diisolasi dari pasien monkeypox dengan dan tanpa riwayat perjalanan internasional untuk memahami hubungan filogenetik dan evolusi genom virus yang dapat berkontribusi pada tingkat transmisi yang lebih tinggi.
Latar belakang
Virus monkeypox, seperti virus Variola penyebab cacar,
adalah spesies Orthopoxvirus yang termasuk dalam famili Poxviridae. Ini adalah
virus double-stranded deoxyribonucleic acid (DNA) dengan genom yang mengkodekan
sekitar 190 gen.
Kasus cacar monyet pertama yang diketahui manusia terjadi
pada tahun 1970 di Republik Demokratik Kongo, yang menyebar ke negara-negara di
Afrika barat dan tengah, dengan wabah berikutnya di Amerika Serikat (AS) pada
tahun 2003, dan Inggris Raya (Inggris Raya) pada tahun 2018. Studi genom yang
dilakukan oleh World Health Organization (WHO) mengenali dua clade utama -
Clade I (clade Cekungan Kongo) dan Clade II (clade Afrika Barat), dengan dua
subclade di Clade II. Wabah cacar monyet global tahun 2022 telah dikaitkan
dengan Clade IIb.
Virus dari keluarga Poxviridae telah menunjukkan frekuensi
rekombinasi antar dan intra-spesies yang tinggi. Clades I dan II memiliki jarak
genetik 900 bp. Perubahan genetik dapat membuat transmisi dan infeksi
menguntungkan virus. Oleh karena itu, seluruh analisis genom dari strain virus
monkeypox yang beredar sangat penting dalam pengawasan penyakit.
Tentang studi
Penelitian ini mengumpulkan sampel dari swab nasofaring dan
orofaringeal, krusta lesi, dan cairan dari 96 orang yang diduga menderita cacar
monyet. Kasus positif diidentifikasi berdasarkan real-time polymerase chain
reaction (PCR) menggunakan primer spesifik monkeypox.
Kumpulan sampel terakhir termasuk lima sampel dari Kerala
(dengan riwayat perjalanan internasional) dan lima dari Delhi (tanpa riwayat
perjalanan internasional baru-baru ini). Sampel negatif disaring lebih lanjut
untuk enterovirus dan virus Varicella zoster.
Sekuensing generasi berikutnya digunakan untuk karakterisasi
genom dari sampel positif. Selain itu, analisis filogenetik dilakukan pada
dataset akhir yang terdiri dari sekuens genom yang dihasilkan dalam penelitian
ini dan 75 sekuens dari database NCBI (National Center for Biotechnology
Information) dan GISAID (Global Initiative on Sharing Avian Influenza Data)
yang dihasilkan pra- dan wabah cacar monyet pasca 2022.
Hasil studi
Studi ini mengambil 90% hingga 99% genom cacar monyet dari
kerak lesi dan sampel cairan dari semua kasus positif. Pohon filogenetik
menempatkan 10 sampel dari India dalam silsilah Clade IIb A.2, bersama dengan
delapan sampel lainnya dari AS, Thailand, dan Inggris.
Analisis berbasis genom membagi garis keturunan A.2 menjadi
tiga sub-cluster, dengan tujuh sekuens (lima dari Kerala, dan dua dari Delhi)
bercabang dengan galur UK-2022 OP331335.1 dan USA-2022 ON674051,1 dan membentuk
satu sub-klaster. Mutasi yang menentukan garis keturunan pada posisi C 179537
T, C 25072 T, dan A 140492 C selanjutnya mengklasifikasikan lima urutan dari
Kerala sebagai sub-garis keturunan A.2.1.
Tiga sekuens yang tersisa dari Delhi bercabang dengan strain
USA-2022 ON675438.1 dan membentuk sub-cluster kedua. Sub-cluster ketiga terdiri
dari sekuens dari strain AS dan Inggris lainnya serta sampel dari Thailand.
Penelitian telah menghubungkan evolusi jangka pendek dari
virus monkeypox dengan mutasi pada gen apolipoprotein B editing complex
(APOBEC3) yang menghasilkan aktivitas cytidine deaminase. Analisis genom dari
penelitian ini mengungkapkan 13 mutasi APOBEC3 baru dan 16 single nucleotide
polymorphisms (SNPs), yang penulis yakini sebagai perubahan yang menentukan
garis keturunan dalam garis keturunan A.2. Studi ini juga mengidentifikasi 25
mutasi APOBEC3 tambahan pada jenis cacar monyet yang beredar di India.
Urutan cacar monyet dari India berbeda dari garis keturunan
sebelumnya, seperti B.1 dari negara-negara Eropa seperti Jerman, Italia, dan
Prancis, dan garis keturunan A.1 2017-2018 dari Nigeria, Singapura, dan Israel.
Kesimpulan
Sebagai rangkuman, penelitian ini melaporkan 13 mutasi baru
pada gen APOBEC3 virus monkeypox yang berpotensi terlibat dalam evolusi virus.
Selain itu, hasilnya juga menemukan 16 SNP baru yang, bersama dengan mutasi
APOBEC3, menghasilkan sampel dari India yang membentuk garis keturunan A.2.1.
Para penulis percaya bahwa mutasi pada gen Orthopoxvirus dikaitkan dengan
peningkatan virulensi melalui penekanan kekebalan pada inang.
Selanjutnya, gen di wilayah terminal virus monkeypox dan
spesies Orthopoxvirus lainnya bertanggung jawab atas adaptasi dan transmisi
inang. Oleh karena itu, studi luas genom untuk memahami peran gen-gen ini dan
pemantauan mutasi yang muncul sangat penting untuk menahan penyebaran cacar
monyet.
*Pemberitahuan Penting
bioRxiv menerbitkan laporan ilmiah awal yang tidak ditinjau
oleh rekan sejawat dan, oleh karena itu, tidak boleh dianggap sebagai
konklusif, memandu praktik klinis/perilaku yang berhubungan dengan kesehatan,
atau diperlakukan sebagai informasi yang mapan.
Journal reference:
Genome characterization of monkeypox cases detected in
India: Identification of three sub clusters among A.2 lineage: Anita M Shete,
Pragya D Yadav, Abhinendra Kumar, Savita Patil, Deepak Y Patil, Yash Joshi,
Triparna Majumdar, Vineet Relhan, Rima R Sahay, Meenakshy Vasu, Pranita
Gawande, Ajay Verma, Arbind Kumar, Shivram Dhakad, Anukumar Bala Krishnan,
Shubin Chenayil, Suresh Kumar, and Priya Abraham. bioRxiv. 2022. DOI:
https://doi.org/10.1101/2022.09.16.507742, https://www.biorxiv.org/content/10.1101/2022.09.16.507742v1

.jpg)
.jpg)
.jpg)





No comments